Bioterapéuticos: ¿Qué hacemos ahora?
- Detalles
- Categoría: BIOVIA
- Visto: 1946
La pregunta de qué debemos hacer a continuación ha desafiado al mundo del descubrimiento de fármacos durante décadas. Durante años, los métodos computacionales para el diseño de fármacos de moléculas pequeñas han ofrecido numerosos algoritmos y metodologías para ayudar a generar nuevas ideas y guiar el proceso iterativo de diseño y optimización de leads. Para un objetivo farmacológico en particular, estos métodos ayudan a identificar candidatos de alta calidad que eventualmente pueden avanzar al desarrollo clínico con menos experimentos y tiempo en el laboratorio. Desde los primeros días de la química combinatoria y el reemplazo bioisostérico hasta el diseño basado en ligandos, fragmentos y estructuras, ha habido muchas herramientas que aprovechan numerosos algoritmos que se adaptan a las limitaciones y criterios de diseño de su proyecto. Más recientemente, los algoritmos de IA y aprendizaje automático han sido populares para permitir a los investigadores explorar rápidamente más ideas en el espacio químico y proponer estructuras novedosas que un químico medicinal puede no haber considerado probar cuando busca nuevos medicamentos.
Hasta hace poco, las herramientas de diseño computacional para la bioterapéutica parecían requerir más experiencia, y ser más escasas y específicas de la aplicación en comparación con las herramientas que existen para la terapéutica de moléculas pequeñas. Por supuesto, existen algoritmos de diseño computacional disponibles, como el modelado de homología, el acoplamiento proteína-proteína y la mutagénesis de escaneo combinatorio para el modelado general de proteínas y el diseño de aglutinantes, que se utilizan en el descubrimiento y la optimización de pistas bioterapéuticas. Para el diseño de ciertos tipos de terapias biológicas, como los anticuerpos monoclonales, existen métodos como la maduración por afinidad, la humanización y los algoritmos de predicción de inmunogenicidad. Sin embargo, para ayudar a responder directamente qué variación de nuestro producto bioterapéutico deberíamos hacer y probar a continuación, dos métodos recientes de IA, RFDiffusion y ProteinMPNN, han cambiado totalmente la naturaleza del descubrimiento bioterapéutico. Estas herramientas tienen el potencial de cambiar la forma en que diseñamos bioterapéuticos al ayudar a identificar nuevos candidatos que los biólogos computacionales y moleculares pueden no haber considerado.
RFDiffusion es un algoritmo de IA generativa de última generación que puede "difundir" una colección de aminoácidos en una estructura de proteína. El proceso de difusión comienza con una colección aleatoria y ruidosa de átomos y, a través de una serie de refinamientos controlados, el algoritmo realiza ajustes en la estructura para reducir el ruido y acercarse a una estructura de proteína biológicamente realista y funcional. Una analogía común para el proceso de difusión es revelar una foto a partir de una imagen borrosa; Los pasos de procesamiento iterativos pueden tomar una imagen granulada inicial y refinar el detalle y la claridad para producir una imagen clara final.
RFDiffusion se puede utilizar para una serie de diferentes desafíos de diseño bioterapéutico, como la ingeniería de un producto biológico que pueda unirse a una proteína viral para neutralizar el virus. Con estructuras de anticuerpos u otros sistemas proteína-proteína, RFDiffusion se puede utilizar para diseñar nuevos andamios de proteínas que puedan mejorar las afinidades de unión o mejorar la estabilidad de los socios de unión. RFDiffusion también se puede utilizar para generar terapias enzimáticas que pueden descomponer un sustrato específico para tratar trastornos metabólicos. Más allá de la bioterapia, RFDiffusion tiene potencial para ayudar a diseñar proteínas para aplicaciones industriales y biotecnológicas, como la fabricación de enzimas que catalizan reacciones químicas específicas o proteínas que se adaptan a condiciones muy específicas, como baja o alta temperatura, pH, etc.
ProteinMPNN es una red neuronal de última generación que puede predecir una o más secuencias de proteínas probables dada una estructura proteica. Este algoritmo se ha publicado con éxito en uno de los aspectos más críticos del diseño de secuencias de proteínas: generar secuencias que se pliegan en una proteína/péptido estable con propensión a cristalizar, lo que facilita la determinación de la estructura de estas proteínas. ProteinMPNN se puede utilizar junto con RFDiffusion para generar nuevos diseños de proteínas, como nuevas enzimas o anticuerpos, que se pueden evaluar más a fondo para determinar las propiedades deseadas, como la estabilidad, la actividad, la afinidad y la especificidad. Uno de los puntos fuertes de ProteinMPNN es su capacidad para generar múltiples variantes de secuencia. Esta capacidad es invaluable, ya que las diferentes variantes brindan más opciones para probar e identificar candidatos con el mejor rendimiento en términos de eficacia, seguridad y capacidad de fabricación. De igual manera, estas variantes también proporcionan pistas alternativas cuando los candidatos encuentran problemas imprevistos en la optimización de proteínas, durante la expresión de proteínas o desafíos de ADMET, como la solubilidad y la inmunogenicidad.
Juntos, RFDiffusion y ProteinMPNN amplían significativamente el espacio biológico que se puede explorar in silico antes de que los biólogos tengan que comprometerse con una experimentación física costosa y que requiere mucho tiempo. Tienen el potencial de abrir vías interesantes para flujos de trabajo más inteligentes, basados en modelos y datos, que impulsen la innovación en el diseño bioterapéutico.
En BIOVIA Discovery Studio Simulation, un nuevo protocolo Generate Protein Scaffolds proporciona ahora un fácil acceso a los flujos de trabajo de RFDiffusion, el primero de los cuales es el andamiaje de motivos. Los usuarios pueden comenzar con una parte específica de una proteína existente (el motivo) y diseñar un andamio de proteína completamente nuevo que incorpore este motivo. Este enfoque permite un control preciso sobre las regiones funcionales de la proteína, así como un control sobre el diseño del andamiaje de la proteína, a través de diferentes pesos de modelo que se adaptan a proteínas y complejos particulares.
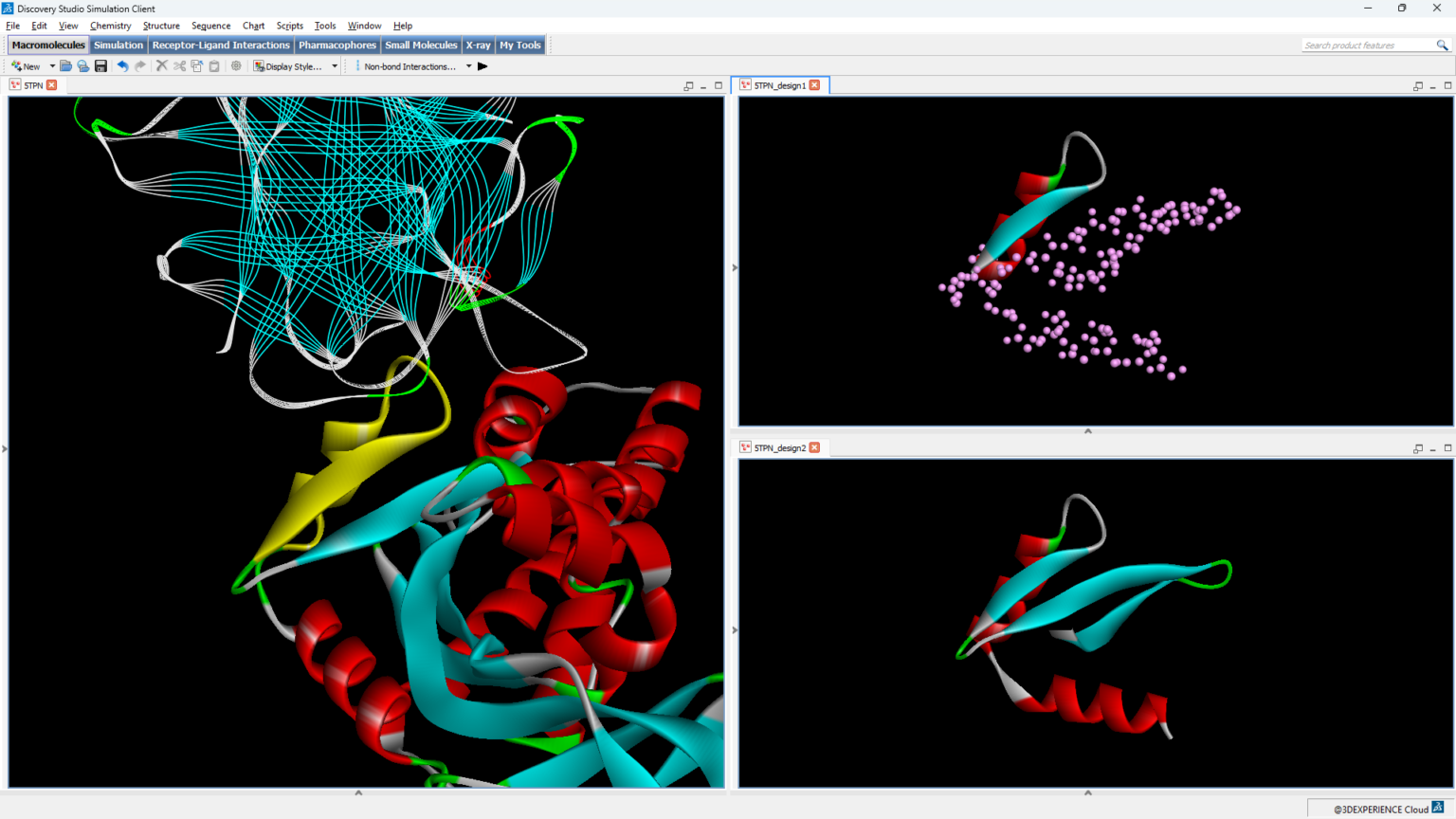 Figura 1- Los usuarios de Discovery Studio Simulation ahora tienen acceso al andamiaje de motivos con RFDiffusion.
Figura 1- Los usuarios de Discovery Studio Simulation ahora tienen acceso al andamiaje de motivos con RFDiffusion.
Un segundo protocolo nuevo, Generate Protein Sequences, permite a los usuarios acceder no solo a ProteinMPNN, donde pueden definir fácilmente los residuos de la secuencia para el diseño, sino también a los modelos LigandMPNN y SolubleMPNN. LigandMPNN es una extensión de ProteinMPNN que es capaz de considerar ligandos de proteínas, moléculas pequeñas, ácidos nucleicos e iones metálicos como contexto adicional para el diseño de secuencias, con el potencial de mejorar las propiedades químicas de las secuencias diseñadas. SolubleMPNN podría ser un mejor modelo para usar cuando la solubilidad de las proteínas es parte de sus criterios de diseño. Los usuarios pueden determinar el grado de diversidad de secuencias y la confianza deseada, como parte del diseño generativo, y tienen la capacidad de controlar el sesgo de aminoácidos particulares.
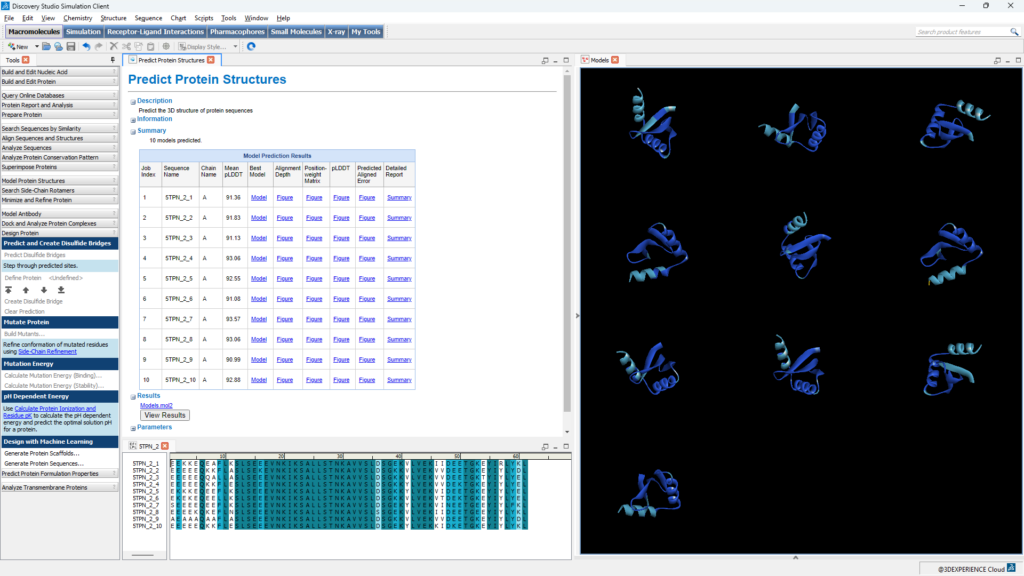 Figura 2- Los usuarios de Discovery Studio Simulation ahora pueden generar nuevas secuencias utilizando modelos ProteinMPNN y usar AlphaFold / OpenFold para generar sus estructuras 3D para aplicaciones posteriores.
Figura 2- Los usuarios de Discovery Studio Simulation ahora pueden generar nuevas secuencias utilizando modelos ProteinMPNN y usar AlphaFold / OpenFold para generar sus estructuras 3D para aplicaciones posteriores.
Estas dos nuevas e importantes mejoras son interesantes adiciones a las herramientas bioterapéuticas y de diseño de proteínas en Discovery Studio Simulation en el 3DEXPERIENCIA® en la nube, que ya incluye la predicción de estructuras AlphaFold y OpenFold AI. Amplían el arsenal cada vez mayor de potentes herramientas de IA para modeladores moleculares y biólogos para ayudar a responder a la pregunta de "qué hacer y probar a continuación" y acelerar el diseño racional de productos biológicos. En combinación con los métodos basados en la física existentes en Discovery Studio Simulation, los usuarios pueden explorar rápidamente muchas más posibilidades in silico antes de llegar al puñado final de candidatos que están listos para convertirse en un bioterapéutico comercial exitoso o un biológico para su uso en la agricultura, la alimentación y las bebidas, o las industrias medioambientales.
Los Premios Nobel de Química y Física de este año celebran cómo la IA está ampliando los límites de la investigación científica. John J. Hopfield y Geoffrey E. Hinton fueron galardonados con el Premio Nobel de Física por sus descubrimientos fundamentales en el aprendizaje automático con redes neuronales artificiales, mientras que David Baker, Demis Hassabis y John Jumper recibieron el Premio Nobel de Química por sus avances en el diseño computacional de proteínas y la predicción de su estructura.
En BIOVIA, estamos orgullosos de formar parte de esta revolución de la IA. Al integrar AlphaFold2, OpenFold, RFDiffusion y la familia de modelos ProteinMPNN en nuestra plataforma, dotamos a los investigadores de herramientas de vanguardia para la predicción de la estructura y el diseño de proteínas.
Vea el vídeo para obtener más información sobre cómo Discovery Studio Simulation ahora ayuda a los usuarios a generar productos biológicos novedosos con los modelos RFDiffusion y LigandMPNN.
Hasta hace poco, las herramientas de diseño computacional para la bioterapéutica parecían requerir más experiencia, y ser más escasas y específicas de la aplicación en comparación con las herramientas que existen para la terapéutica de moléculas pequeñas. Por supuesto, existen algoritmos de diseño computacional disponibles, como el modelado de homología, el acoplamiento proteína-proteína y la mutagénesis de escaneo combinatorio para el modelado general de proteínas y el diseño de aglutinantes, que se utilizan en el descubrimiento y la optimización de pistas bioterapéuticas. Para el diseño de ciertos tipos de terapias biológicas, como los anticuerpos monoclonales, existen métodos como la maduración por afinidad, la humanización y los algoritmos de predicción de inmunogenicidad. Sin embargo, para ayudar a responder directamente qué variación de nuestro producto bioterapéutico deberíamos hacer y probar a continuación, dos métodos recientes de IA, RFDiffusion y ProteinMPNN, han cambiado totalmente la naturaleza del descubrimiento bioterapéutico. Estas herramientas tienen el potencial de cambiar la forma en que diseñamos bioterapéuticos al ayudar a identificar nuevos candidatos que los biólogos computacionales y moleculares pueden no haber considerado.
Generación de proteínas con IA: RFDiffusion y ProteinMPNN
RFDiffusion es un algoritmo de IA generativa de última generación que puede "difundir" una colección de aminoácidos en una estructura de proteína. El proceso de difusión comienza con una colección aleatoria y ruidosa de átomos y, a través de una serie de refinamientos controlados, el algoritmo realiza ajustes en la estructura para reducir el ruido y acercarse a una estructura de proteína biológicamente realista y funcional. Una analogía común para el proceso de difusión es revelar una foto a partir de una imagen borrosa; Los pasos de procesamiento iterativos pueden tomar una imagen granulada inicial y refinar el detalle y la claridad para producir una imagen clara final.
RFDiffusion se puede utilizar para una serie de diferentes desafíos de diseño bioterapéutico, como la ingeniería de un producto biológico que pueda unirse a una proteína viral para neutralizar el virus. Con estructuras de anticuerpos u otros sistemas proteína-proteína, RFDiffusion se puede utilizar para diseñar nuevos andamios de proteínas que puedan mejorar las afinidades de unión o mejorar la estabilidad de los socios de unión. RFDiffusion también se puede utilizar para generar terapias enzimáticas que pueden descomponer un sustrato específico para tratar trastornos metabólicos. Más allá de la bioterapia, RFDiffusion tiene potencial para ayudar a diseñar proteínas para aplicaciones industriales y biotecnológicas, como la fabricación de enzimas que catalizan reacciones químicas específicas o proteínas que se adaptan a condiciones muy específicas, como baja o alta temperatura, pH, etc.
ProteinMPNN es una red neuronal de última generación que puede predecir una o más secuencias de proteínas probables dada una estructura proteica. Este algoritmo se ha publicado con éxito en uno de los aspectos más críticos del diseño de secuencias de proteínas: generar secuencias que se pliegan en una proteína/péptido estable con propensión a cristalizar, lo que facilita la determinación de la estructura de estas proteínas. ProteinMPNN se puede utilizar junto con RFDiffusion para generar nuevos diseños de proteínas, como nuevas enzimas o anticuerpos, que se pueden evaluar más a fondo para determinar las propiedades deseadas, como la estabilidad, la actividad, la afinidad y la especificidad. Uno de los puntos fuertes de ProteinMPNN es su capacidad para generar múltiples variantes de secuencia. Esta capacidad es invaluable, ya que las diferentes variantes brindan más opciones para probar e identificar candidatos con el mejor rendimiento en términos de eficacia, seguridad y capacidad de fabricación. De igual manera, estas variantes también proporcionan pistas alternativas cuando los candidatos encuentran problemas imprevistos en la optimización de proteínas, durante la expresión de proteínas o desafíos de ADMET, como la solubilidad y la inmunogenicidad.
Juntos, RFDiffusion y ProteinMPNN amplían significativamente el espacio biológico que se puede explorar in silico antes de que los biólogos tengan que comprometerse con una experimentación física costosa y que requiere mucho tiempo. Tienen el potencial de abrir vías interesantes para flujos de trabajo más inteligentes, basados en modelos y datos, que impulsen la innovación en el diseño bioterapéutico.
Generación de proteínas con RFDiffusion y ProteinMPNN en la simulación de Discovery Studio
En BIOVIA Discovery Studio Simulation, un nuevo protocolo Generate Protein Scaffolds proporciona ahora un fácil acceso a los flujos de trabajo de RFDiffusion, el primero de los cuales es el andamiaje de motivos. Los usuarios pueden comenzar con una parte específica de una proteína existente (el motivo) y diseñar un andamio de proteína completamente nuevo que incorpore este motivo. Este enfoque permite un control preciso sobre las regiones funcionales de la proteína, así como un control sobre el diseño del andamiaje de la proteína, a través de diferentes pesos de modelo que se adaptan a proteínas y complejos particulares.
 Figura 1- Los usuarios de Discovery Studio Simulation ahora tienen acceso al andamiaje de motivos con RFDiffusion.
Figura 1- Los usuarios de Discovery Studio Simulation ahora tienen acceso al andamiaje de motivos con RFDiffusion.Un segundo protocolo nuevo, Generate Protein Sequences, permite a los usuarios acceder no solo a ProteinMPNN, donde pueden definir fácilmente los residuos de la secuencia para el diseño, sino también a los modelos LigandMPNN y SolubleMPNN. LigandMPNN es una extensión de ProteinMPNN que es capaz de considerar ligandos de proteínas, moléculas pequeñas, ácidos nucleicos e iones metálicos como contexto adicional para el diseño de secuencias, con el potencial de mejorar las propiedades químicas de las secuencias diseñadas. SolubleMPNN podría ser un mejor modelo para usar cuando la solubilidad de las proteínas es parte de sus criterios de diseño. Los usuarios pueden determinar el grado de diversidad de secuencias y la confianza deseada, como parte del diseño generativo, y tienen la capacidad de controlar el sesgo de aminoácidos particulares.
 Figura 2- Los usuarios de Discovery Studio Simulation ahora pueden generar nuevas secuencias utilizando modelos ProteinMPNN y usar AlphaFold / OpenFold para generar sus estructuras 3D para aplicaciones posteriores.
Figura 2- Los usuarios de Discovery Studio Simulation ahora pueden generar nuevas secuencias utilizando modelos ProteinMPNN y usar AlphaFold / OpenFold para generar sus estructuras 3D para aplicaciones posteriores.Estas dos nuevas e importantes mejoras son interesantes adiciones a las herramientas bioterapéuticas y de diseño de proteínas en Discovery Studio Simulation en el 3DEXPERIENCIA® en la nube, que ya incluye la predicción de estructuras AlphaFold y OpenFold AI. Amplían el arsenal cada vez mayor de potentes herramientas de IA para modeladores moleculares y biólogos para ayudar a responder a la pregunta de "qué hacer y probar a continuación" y acelerar el diseño racional de productos biológicos. En combinación con los métodos basados en la física existentes en Discovery Studio Simulation, los usuarios pueden explorar rápidamente muchas más posibilidades in silico antes de llegar al puñado final de candidatos que están listos para convertirse en un bioterapéutico comercial exitoso o un biológico para su uso en la agricultura, la alimentación y las bebidas, o las industrias medioambientales.
Premios Nobel de Química y Física
Los Premios Nobel de Química y Física de este año celebran cómo la IA está ampliando los límites de la investigación científica. John J. Hopfield y Geoffrey E. Hinton fueron galardonados con el Premio Nobel de Física por sus descubrimientos fundamentales en el aprendizaje automático con redes neuronales artificiales, mientras que David Baker, Demis Hassabis y John Jumper recibieron el Premio Nobel de Química por sus avances en el diseño computacional de proteínas y la predicción de su estructura.
En BIOVIA, estamos orgullosos de formar parte de esta revolución de la IA. Al integrar AlphaFold2, OpenFold, RFDiffusion y la familia de modelos ProteinMPNN en nuestra plataforma, dotamos a los investigadores de herramientas de vanguardia para la predicción de la estructura y el diseño de proteínas.
Vea el vídeo para obtener más información sobre cómo Discovery Studio Simulation ahora ayuda a los usuarios a generar productos biológicos novedosos con los modelos RFDiffusion y LigandMPNN.

