BIOVIA Discovery Studio frente COVID-19: modos blandos
- Detalles
- Categoría: BIOVIA
- Visto: 3552
El investigador del del (CSIC), , ha estado utilizando BIOVIA Discovery Studio para analizar los modos normales de la proteína S del virus SARS-CoV-2, como estrategia en la búsqueda de posibles agentes que generen inmunidad/tratamiento frente al COVID-19.
El estudio de los modos normales de menor frecuencia (modos blandos) es útil para obtener estimaciones cualitativas de muchas propiedades funcionales de las macromoléculas biológicas. Por lo tanto, la ubicación de los modos blandos ayuda a describir aquellas regiones proteicas donde hay un movimiento de dominio significativo y a extraer la información transmitida por las vibraciones clave.
La protección contra el SARS-CoV-2 actual y otros futuros virus pandémicos con anticuerpos o fármacos candidatos podría reconocerse en sílice siguiendo el comportamiento previamente aprendido de sistemas similares1,2 como se muestra en el siguiente gráfico:
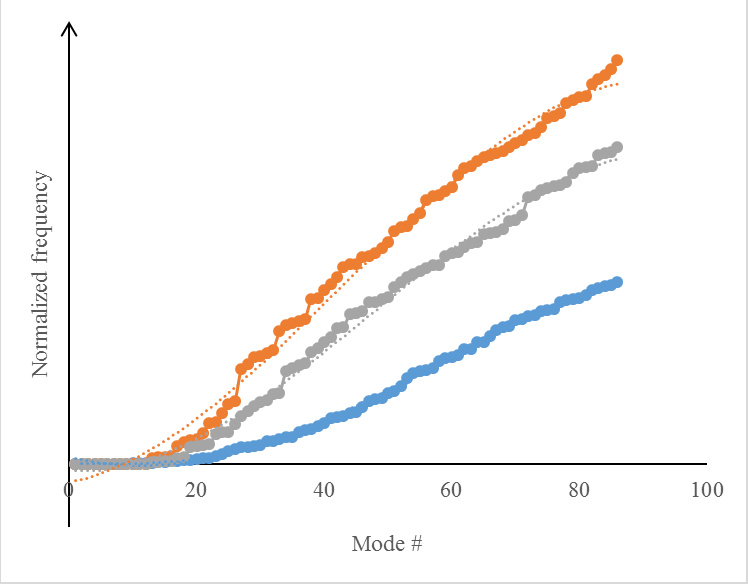
En lugar de tomar la estructura de proteína aislada para el análisis de modos blandos, podemos realizar dicho estudio con el par proteína/anticuerpo o proteína/fármaco si buscamos la eficacia de la inmunidad o el tratamiento farmacológico.
1Datos computacionales realizados con las estructuras descritas en Ekiert et al., Reconocimiento de anticuerpos de un epítopo altamente conservado del virus de la influenza: implicaciones para la prevención y la terapia universal. Science, 2009, 324, 246-251. 2Los algoritmos para el análisis computacional se realizaron con Dassault Systèmes BIOVIA, [Discovery Studio Client v18.1100.18065], San Diego: Dassault Systèmes, [2017].
